Outils
Génome de référence
L’état du génome de référence du caribou
| Plateforme de séquençage | Taille du génome (Gbp) | Nombre de scaffolds | N50 (pb) | L90 (Nombre de scaffolds représentant 90% du génome) |
| 10X Genomics | 2,559 | 21 785 | 2 383 988 | 742 |
| PacBio | 2,526 | 6 351 | 506 219 | 5 130 |
| Dovetail* | 2,205 | 4 699 | 11 765 000 | 289 |
| Intégration (toutes les données) | 2,590 | 13 982 | 40 742 917 | 130 |
Les SNPs « Single Nucleotide Polymorphism »
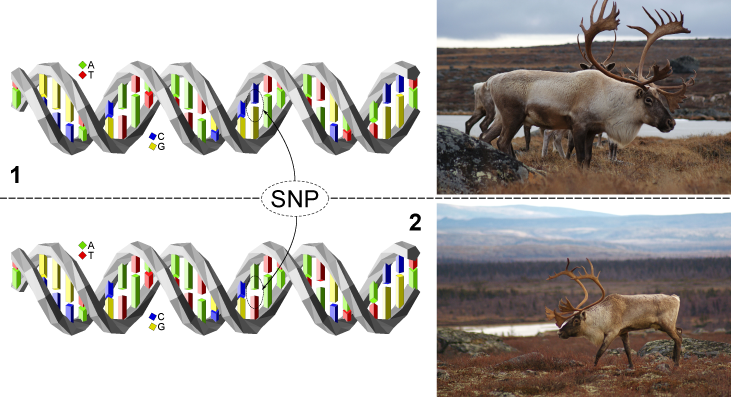
L’ADN est en constitué d’une suite de combinaisons de 4 différents nucléotides représentés par les lettres A, C, G et T. Un SNP désigne un endroit dans la séquence d’ADN où on trouve une lettre différente entre deux brins d’ADN. En d’autres mots, nous sommes tous différents en grande partie à cause de nos différences dans notre séquence d’ADN. Chez les mammifères, le processus reproductif implique un accouplement entre un mâle et une femelle. Ces deux individus étant différents, la séquence d’ADN du père comporte des différences par rapport à la séquence transmise par la mère. Ainsi, nous sommes tous porteurs de deux brins d’ADN qui sont quasi identiques mis à part certaines différences qui nous rendent unique. La nature de ces différences est multiple parce que toutes les modifications possibles et imaginables que nous puissions faire dans une suite de lettres se retrouvent dans tous les génomes. Ainsi, il est possible d’ajouter des lettres, d’en enlever ou de substituer une lettre pour une autre.
Le SNP représente le dernier cas où une lettre est remplacée par une autre. Il est possible de modifier la séquence de plus d’une lettre consécutive mais les modifications associées au terme SNP représente le cas où une seule lettre est modifiée.
Les génomes de mammifères ont une taille assez similaire avoisinant les 3 milliards de lettres. Les SNPs sont relativement fréquents et chaque génome en compte possiblement environ 10 millions si on se fie à la moyenne très grossière de 1 SNP pour chaque 300 lettres. Ce ne sont que des estimations mais ce nombre de SNPs implique qu’ils sont distribués partout dans le génome. Puisque ces différences sont héritées des parents, une description de ces SNPs d’un individu à l’autre permet de décrire son génome et de le comparer aux autres. Ceci permet, entre autres, d’identifier des familles à l’intérieur des populations, d’établir des liens de parenté, de déterminer la variabilité génétique à l’intérieur de chaque groupe de même qu’entre les groupes.
Ces informations sont importantes parce qu’en règle générale, une population se porte bien quand plusieurs familles contribuent à la descendance et plus il y a de diversité génétique, plus la population est en mesure de s’adapter à des changements de toute nature. À l’inverse, une population qui est très homogène est généralement plus vulnérable face aux stress.
La puce à SNPs
La puce à SNPs est simplement une technologie pour interroger un génome de façon à déterminer la lettre trouvée à un endroit précis. Le génotypage est le terme utilisé pour identifier une seule lettre à des centaines voire des centaines de milliers de régions dans un génome. Le principe à la base du génotypage implique en premier lieu de savoir où sont les SNPs à interroger et quelles sont les lettres possibles. Bien qu’il existe différentes variantes technologiques offertes par différents fabricants, le concept général implique d’immobiliser sur une surface solide un minuscule fragment d’ADN correspondant à la séquence de lettres tout juste à côté de l’endroit où se trouve le SNP d’intérêt. L’ADN de l’individu est fragmenté en petits morceaux et par complémentarité de la suite de lettres, le fragment complémentaire à la suite de lettres immobilisée viendra s’apparier pour former le double brin d’ADN. Il suffit ensuite d’ajouter successivement chacune des quatre lettres possibles (A, G, T et C) et de déterminer laquelle a été ajoutée au bout du brin immobilisé. Chacune des quatre lettres porte une molécule fluorescente différente. Selon les couleurs détectées, il est possible de déterminer la lettre qui a été ajoutée. Chaque individu est composé de deux brins d’ADN (un reçu de son père et l’autre de sa mère) ce qui porte à deux le nombre maximal de couleurs potentiellement détectées pour chaque individu pour chaque SNP. La miniaturisation de ce procédé permet maintenant d’interroger près de 1,2 million de SNPs sur une surface de la grandeur d’une lame de microscope (2,5 cm par 7,5 cm). Dans le cadre de l’outil que nous développons pour le caribou/ renne, la puce vise à interroger 60 000 SNPs. Ces derniers sont distribués partout dans le génome de façon à générer de l’information sur toute la longueur de la molécule d’ADN. |  |

Figure du principe général du génotypage
La puce de génotypage est utilisable à partir de l’ADN extrait de diverses sources telles que :
- Échantillon sanguin
- Biopsie de peau
- Échantillon de poils avec la racine
- Mucus prélevé sur les fèces
- Tous autres tissus de l’animal
BLAST « Basic Local Alignment Search Tool »
Le Blast permet de chercher une séquence spécifique sur le génome de référence. En gros, l’algorithme cherche des séquences qui sont identiques ou partiellement identiques entre la séquence d’ADN de la requête et les séquences de nucléotides composant le génome du caribou. Les applications de cet outils sont multiples et permet entre autres de:
- Déterminer si la séquence d’intérêt fait partie du génome de référence
- Déterminer si la séquence d’intérêt est situé près d’un gène connu
- Identifier des variations dans la séquence de l’ADN qui pourraient éventuellement être associées à des caractéristiques trouvés sur les animaux (ex. caractéristiques physiques ou comportementales)
À venir !
Cet outil est présentement en construction et sera disponible sous peu.
Genome Browser
Le « Genome browser » permet de surfer sur le génome de référence afin de visualiser certaines régions d’intérêt. Il est possible de faire une recherche en utilisant les coordonnées génomiques ou un nom de gène. Un peu comme une carte de type « Google Map », il est possible de visualiser une région dans son ensemble ou d’agrandir un segment précis pour visualiser les éléments connus du génome. Il est aussi possible d’ajouter des informations sur le génome de référence en utilisant les coordonnées génomiques.
À venir !
Cet outil est présentement en construction et sera disponible sous peu.
CARLA « CARibou LIMS and Analysis »
CARLA est un portail permettant aux utilisateurs d’analyser leurs résultats de génotypage. L’accès est sécurisé par un mot de passe pour chaque usager de façon à permettre l’accès à un environnement indépendant et sécurisé pour chacun. La partie LIMS de CARLA (« Laboratory Information Management System ») permet de recueillir des informations de base sur les échantillons (ex. type d’échantillon, sexe de l’animal, coordonnées d’échantillonnage, etc.). L’environnement de chaque usager est privé mais il est possible de soumettre des échantillons au domaine public afin de bâtir une base de données en temps réel sur la génomique du caribou. La partie d’analyse de CARLA permet de choisir des groupes d’échantillons et de les comparer afin d’en tirer des métriques de base (ex. regroupements non-supervisés, mesure du coefficient de consanguinité génomique, etc.).
À venir !
Cet outil est présentement en construction et sera disponible sous peu.